R/Конверсия биграфа в монограф
Материал из Letopisi.Ru — «Время вернуться домой»
(Различия между версиями)
(→Граф статей или групп) |
|||
| (не показаны 4 промежуточные версии 1 участника) | |||
| Строка 12: | Строка 12: | ||
g2 <- data.frame(User = paste("U",ghist[,1],sep=":" ) , Page = paste("P",ghist[,2],sep=":") ) # если мы хотим добавим указания на страницы и участников | g2 <- data.frame(User = paste("U",ghist[,1],sep=":" ) , Page = paste("P",ghist[,2],sep=":") ) # если мы хотим добавим указания на страницы и участников | ||
| − | g2 <- data.frame(User = ghist[,1] , Page = ghist[,2] ) # | + | g2 <- data.frame(User = ghist[,1] , Page = ghist[,2] ) # |
| + | |||
| + | И с этим графом - [[биграф]]ом работаем. | ||
| + | |||
| + | |||
| + | ==== bipartite.projection ==== | ||
| + | |||
| + | rm(list=ls()) | ||
| + | df <- data.frame(perpetrator=c("A","A","B","C","D","A","E","A","F","G"), target=c("a","a","a","b","c","d","e","a","f","f")) | ||
| + | net <- graph.edgelist(as.matrix(df)) | ||
| + | V(net)$type <- bipartite.mapping(net)$type | ||
| + | par(mfrow=c(1,2),mar=c(0,1,1,1)) | ||
| + | plot(net, main="Full Network",edge.arrow.size=0.5) | ||
| + | plot(bipartite.projection(net)$proj2,main="Affilitaton Network") | ||
=== Превращение из биграфа в one-mode === | === Превращение из биграфа в one-mode === | ||
| Строка 44: | Строка 57: | ||
gn <- graph_from_adjacency_matrix(Acol, mode = "undirected", diag = TRUE) | gn <- graph_from_adjacency_matrix(Acol, mode = "undirected", diag = TRUE) | ||
gn <- graph_from_adjacency_matrix(Acol, mode = c("directed"), diag = TRUE) | gn <- graph_from_adjacency_matrix(Acol, mode = c("directed"), diag = TRUE) | ||
| + | |||
| + | и еще лучше с учетом веса: | ||
| + | |||
| + | gm <- graph_from_adjacency_matrix(Acol, weighted=T, mode = "undirected") | ||
| + | gm <- simplify(gm) | ||
| + | E(gm)$label = E(gm)$weight # указываем вес на ребрах | ||
| + | set.seed(10) | ||
Now we need to tansform the graph so that multiple edges become an attribute ( E(g)$weight ) of each unique edge: | Now we need to tansform the graph so that multiple edges become an attribute ( E(g)$weight ) of each unique edge: | ||
| Строка 62: | Строка 82: | ||
[[Файл:Eduwn.png]] | [[Файл:Eduwn.png]] | ||
| − | + | ||
| − | Ссылки: | + | === Ссылки: === |
* http://www.r-bloggers.com/working-with-bipartiteaffiliation-network-data-in-r/ | * http://www.r-bloggers.com/working-with-bipartiteaffiliation-network-data-in-r/ | ||
* http://www.r-bloggers.com/working-with-bipartiteaffiliation-network-data-in-r/ - Working with Bipartite/Affiliation Network Data in R (Solomon) | * http://www.r-bloggers.com/working-with-bipartiteaffiliation-network-data-in-r/ - Working with Bipartite/Affiliation Network Data in R (Solomon) | ||
** Data can often be usefully conceptualized in terms affiliations between people (or other key data entities). It might be useful analyze common group membership, common purchasing decisions, or common patterns of behavior.- готовый хороший текст + лабораторные работы на стыке [[R]] + [[SoNIA]] | ** Data can often be usefully conceptualized in terms affiliations between people (or other key data entities). It might be useful analyze common group membership, common purchasing decisions, or common patterns of behavior.- готовый хороший текст + лабораторные работы на стыке [[R]] + [[SoNIA]] | ||
| + | * https://www.r-bloggers.com/collapsing-a-bipartite-co-occurrence-network/ | ||
| + | ** http://f.briatte.org/r/turning-keywords-into-a-co-occurrence-network Turning keywords into a co-occurrence network | ||
| + | To convert a two-mode incidence matrix to a one-mode adjacency matrix, one can simply multiply an incidence matrix by its transpose. | ||
| + | |||
| + | n <- m %*% t(m) %>% | ||
| + | graph_from_adjacency_matrix(mode = "undirected", diag = FALSE, weighted = TRUE) | ||
| + | |||
| + | ---- | ||
[[Категория:Учебная аналитика]] | [[Категория:Учебная аналитика]] | ||
| − | [[Категория:R] | + | [[Категория:R]] |
Текущая версия на 15:50, 25 мая 2019
Превращение биграфа в граф = история превращения графа, в котором есть и субъекты - person и объекты
Исходный пример - есть люди и есть группы, к которым они относятся
- obebukhova Литература
- sch27@tagobr.ru Литература
Содержание |
[править] Загружаем исходный файл и переводим
ghist <- read.csv(file.choose(),sep=",", as.is=T, header=T, encoding ="UTF-8") # чтобы сохранить русские значения ghist <- na.omit(ghist ) # если там были пропущенные значения, то удалим их
g2 <- data.frame(User = paste("U",ghist[,1],sep=":" ) , Page = paste("P",ghist[,2],sep=":") ) # если мы хотим добавим указания на страницы и участников
g2 <- data.frame(User = ghist[,1] , Page = ghist[,2] ) #
И с этим графом - биграфом работаем.
[править] bipartite.projection
rm(list=ls())
df <- data.frame(perpetrator=c("A","A","B","C","D","A","E","A","F","G"), target=c("a","a","a","b","c","d","e","a","f","f"))
net <- graph.edgelist(as.matrix(df))
V(net)$type <- bipartite.mapping(net)$type
par(mfrow=c(1,2),mar=c(0,1,1,1))
plot(net, main="Full Network",edge.arrow.size=0.5)
plot(bipartite.projection(net)$proj2,main="Affilitaton Network")
[править] Превращение из биграфа в one-mode
library('Matrix') # пакет для работы с матрицами
A <- spMatrix(nrow=length(unique(g2$User)),
ncol=length(unique(g2$Page)),
i = as.numeric(factor(g2$User)),
j = as.numeric(factor(g2$Page)),
x = rep(1, length(as.numeric(g2$User))) )
row.names(A) <- levels(factor(g2$User))
colnames(A) <- levels(factor(g2$Page))
Arow <- A %*% t(A) ## Это у нас граф участников
Acol <- t(A) %*% A ## А это у нас связи группы
[править] Отдельно посмотрим на граф участников
Скорее всего, он очень большой, так что лучше бы его в output послать
gu <- graph_from_adjacency_matrix(Arow,mode = c("directed"), weighted = NULL, diag = TRUE, add.colnames = NULL, add.rownames = NA) # получаем граф
gu <- simplify(gu) # Упрощаем граф
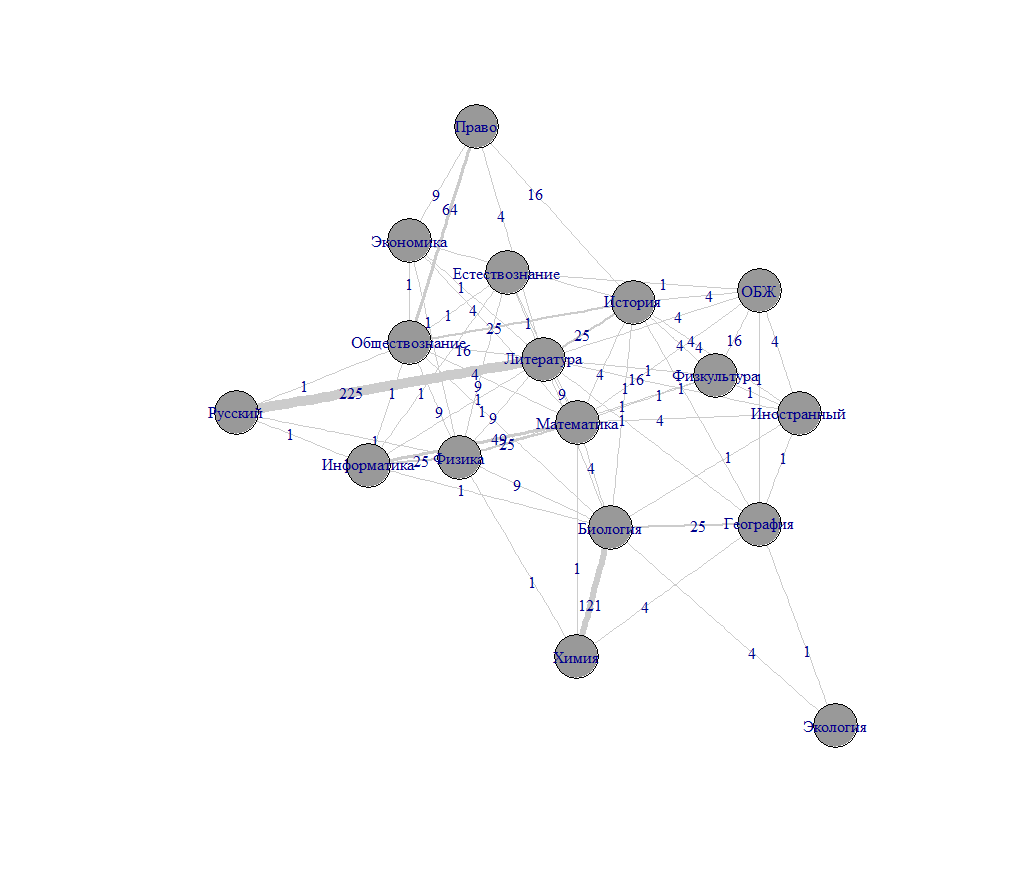
[править] Граф статей или групп
gn <- graph_from_adjacency_matrix(Arow,mode = c("directed"), weighted = NULL, diag = TRUE, add.colnames = NULL, add.rownames = NA) # получаем граф
Или!
gn <- graph_from_adjacency_matrix(Acol, mode = "undirected", diag = TRUE)
gn <- graph_from_adjacency_matrix(Acol, mode = c("directed"), diag = TRUE)
и еще лучше с учетом веса:
gm <- graph_from_adjacency_matrix(Acol, weighted=T, mode = "undirected") gm <- simplify(gm) E(gm)$label = E(gm)$weight # указываем вес на ребрах set.seed(10)
Now we need to tansform the graph so that multiple edges become an attribute ( E(g)$weight ) of each unique edge:
E(gn)$weight <- count.multiple(gn) gn <- simplify(gu) # Упрощаем граф E(gn)$label = E(gn)$weight # Вес ребра будет показан на нем - label plot(gn, layout=layout.fruchterman.reingold, vertex.color="gray60", vertex.size = 15, edge.arrow.size = 0.1, edge.color = "gray80", edge.width = 1+E(gn)$weight/24 )
plot(gn, layout=layout.kamada.kawai, vertex.color="gray60", vertex.size = 25, edge.arrow.size = 0.5, edge.color = "gray80", edge.width = gn$weight )
[править] Ссылки:
- http://www.r-bloggers.com/working-with-bipartiteaffiliation-network-data-in-r/
- http://www.r-bloggers.com/working-with-bipartiteaffiliation-network-data-in-r/ - Working with Bipartite/Affiliation Network Data in R (Solomon)
- https://www.r-bloggers.com/collapsing-a-bipartite-co-occurrence-network/
- http://f.briatte.org/r/turning-keywords-into-a-co-occurrence-network Turning keywords into a co-occurrence network
To convert a two-mode incidence matrix to a one-mode adjacency matrix, one can simply multiply an incidence matrix by its transpose.
n <- m %*% t(m) %>% graph_from_adjacency_matrix(mode = "undirected", diag = FALSE, weighted = TRUE)